Schritt in Richtung schaltbare Gentherapie
Biochemiker der Universität Konstanz entwickeln vereinfachtes Verfahren zur Identifikation von Genschaltern – hohes Anwendungspotenzial für die Gentherapie
Die Gentherapie gilt als aussichtsreiche Methode zur Bekämpfung von Krankheiten: Defekte Gene werden darin in der Zelle gezielt durch intakte Gene ausgetauscht. „Was es in der Gentherapie noch nicht gibt, sind schaltbare Anwendungen“, schildert Prof. Dr. Jörg Hartig, Professor für Biopolymerchemie an der Universität Konstanz. Über sogenannte Genschalter könnten die ausgetauschten Gene gezielt aktiviert und deaktiviert werden, so dass die Gentherapie besser reguliert werden kann. In Zusammenarbeit mit Forscherinnen und Forschern des Unternehmens Boehringer Ingelheim entwickelte das Team von Jörg Hartig nun ein Verfahren, das passgenaue RNA-Genschalter effizient und mit geringem Aufwand identifiziert. Das innovative Verfahren wurde im Wissenschaftsjournal Nature Communications beschrieben.
Zehntausend Kieselsteine und ein Sieb
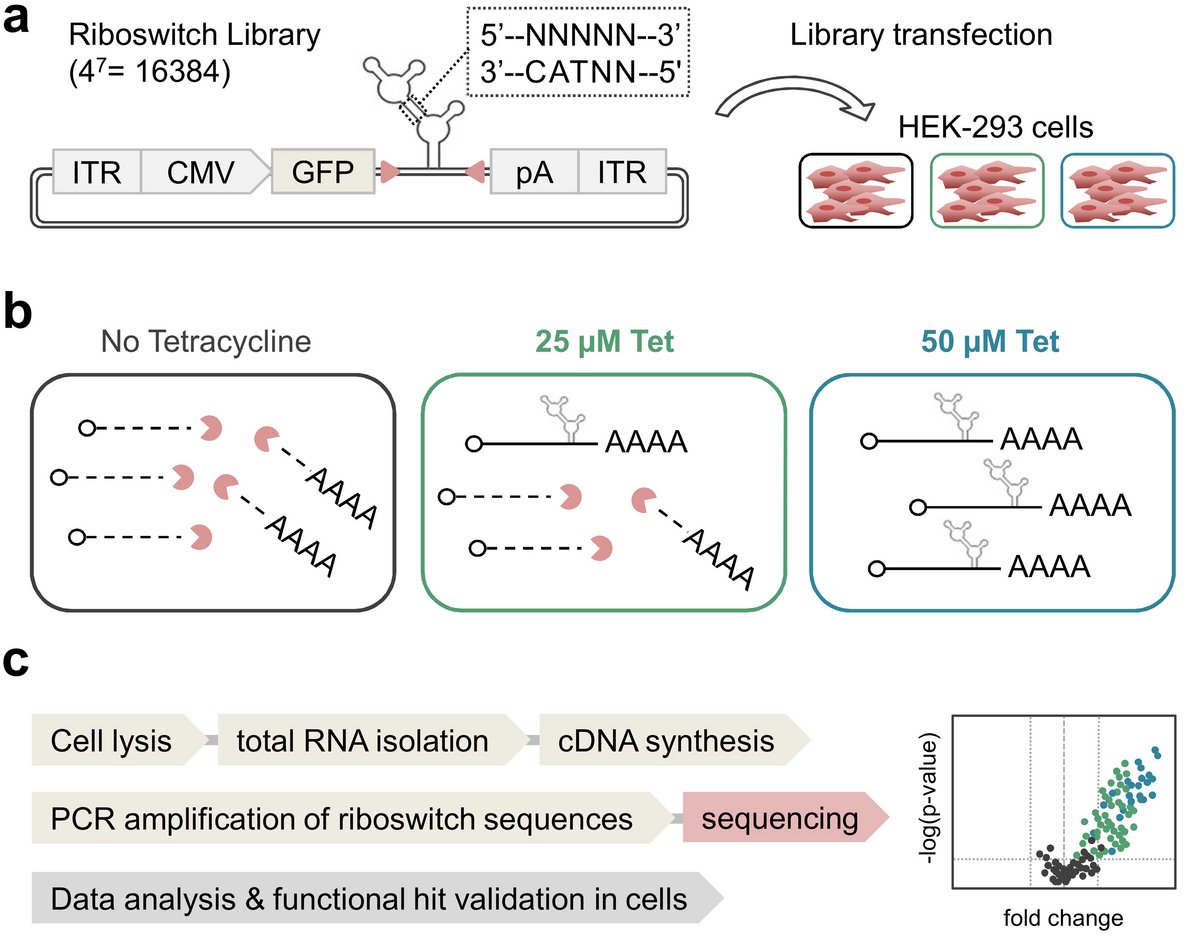
Genschalter aufzuspüren ist bislang ein aufwändiges Verfahren. Aus mehreren zehntausend Ribonukleinsäure-Sequenzen (RNA-Sequenzen) müssen diejenigen gefunden werden, die ein bestimmtes Gen aktivieren oder deaktivieren können. Der Aufwand lässt sich am besten mit einem Vergleich beschreiben: Stellen Sie sich vor, Sie müssten aus zehntausend Kieselsteinen diejenigen heraussortieren, die am kleinsten sind. Ohne Hilfsmittel müssten die Kieselsteine mühselig einzeln durchgesehen oder zumindest in Gruppen aufgeteilt werden. Nun stellen Sie sich vor, Sie hätten ein Sieb zur Hand, durch das alle Kieselsteine gleichzeitig geschüttelt und dadurch nach ihrer Größe sortiert werden können. Dieses „Sieb für Genschalter“ haben Forscher der Arbeitsgruppe von Jörg Hartig und des Unternehmens Boehringer Ingelheim gemeinsam entwickelt. „Anders als in bisherigen Verfahren können wir alle in Frage kommenden RNA-Sequenzen auf einen Schlag untersuchen und nach ihrer Fähigkeit, ein bestimmtes Gen an- oder auszuschalten, staffeln“, erklärt Maike Spöring, Co-Autorin der Studie und Doktorandin in der Arbeitsgruppe von Jörg Hartig.
RNA-basierte Genschalter
„Wir sind spezialisiert auf RNA-basierte Genschalter“, erläutert Jörg Hartig die Grundlage des Verfahrens. Diese Variante der Genschalter ist Teil des RNA-Strangs, der im Zuge der Genexpression genetische Informationen überträgt und in Proteine umsetzt. RNA-Genschalter können gezielt dazu gebracht werden, ihren eigenen RNA-Strang wie eine Schere zu zerschneiden. Ein solcher geschnittener Strang wird im Körper abgebaut; seine genetische Information wird folglich nicht von der Zelle in ein Protein umgesetzt. Über diese Schere lässt sich somit steuern, welche genetischen Informationen der RNA in Proteine umgesetzt werden und welche nicht – wie ein Schalter, der Gene gezielt aktiviert und deaktiviert.
In Hartigs Analyse-Verfahren werden alle Zehntausende von RNA-Sequenzen, die für einen Anwendungszweck als potenzielle Genschalter in Frage kommen, in eine Zellkultur gegeben. Sie werden daraufhin mit dem Molekül, das die Schaltung aktivieren soll, zusammengebracht. Diejenigen RNA-Sequenzen, die damit kompatibel sind, schneiden ihren Strang und werden in der Zellkultur abgebaut. Anhand der Mengenverteilung der verbleibenden RNA-Sequenzen lässt sich ablesen, welche von ihnen als Genschalter funktional sind und wie stark ihre Reaktion ausfällt.
Der Genschalter als Pille
In der Gentherapie würde ein solches schaltbares Gen zunächst in den Körper eingeschleust werden, typischerweise über eine „Genfähre“ wie z. B. ein sogenannter viraler Vektor. In einem zweiten Schritt könnte dann das Molekül, das die Schaltung in Gang setzt, wie ein gewöhnliches Medikament als Pille eingenommen werden. Auf diese Weise ließe sich die Wirkung des ausgetauschten Gens aktivieren und deaktivieren. „Dadurch könnte das Expressionslevel des therapeutischen Proteins genau reguliert werden, also die Dosis, in der das Gen als Protein umgesetzt wird“, gibt Maike Spöring einen Ausblick.
Faktenübersicht:
- Originalpublikation: B. Strobel, M. Spöring, H. Klein, D. Blazevic, W. Rust, S. Sayols Puig, J. S. Hartig, S. Kreuz: High-throughput identification of synthetic riboswitches by barcode-free amplicon-sequencing in human cells. Nature Communications 11, Article number: 714 (2020)
- Konstanzer Forschungsteam um Prof. Dr. Jörg Hartig entwickelte ein Verfahren zur effizienten Identifikation von RNA-basierten Genschaltern. Das Verfahren ermöglicht die Analyse von mehreren zehntausend in Frage kommenden RNA-Sequenzen auf einen Schlag.
- Hohes Anwendungspotenzial im Bereich der Gentherapie.
- Analyse basiert auf sogenannten Next-Generation-RNA-Sequencing-Methoden.
- Prof. Dr. Jörg Hartig ist Professor für Biopolymerchemie an der Universität Konstanz.
- Die Forschung erfolgte in Kooperation mit dem Unternehmen Boehringer Ingelheim und wurde von ihm finanziell gefördert.