Entwicklung des Immunsystems
Wissenschaftler der Universität Konstanz identifizieren Wettstreit zwischen menschlichem Immunsystem und bakteriellen Krankheitserregern
Zellbiologen der Universität Konstanz publizieren in der Fachzeitschrift „Current Biology“ neue Erkenntnisse über die rasante evolutionäre Anpassung des menschlichen Immunsystems: Umfangreiche Genomsequenzierungen ermöglichen Aussagen über die Entwicklung des Rezeptormoleküls CEACAM3 zur Erkennung von bakteriellen Krankheitserregern.
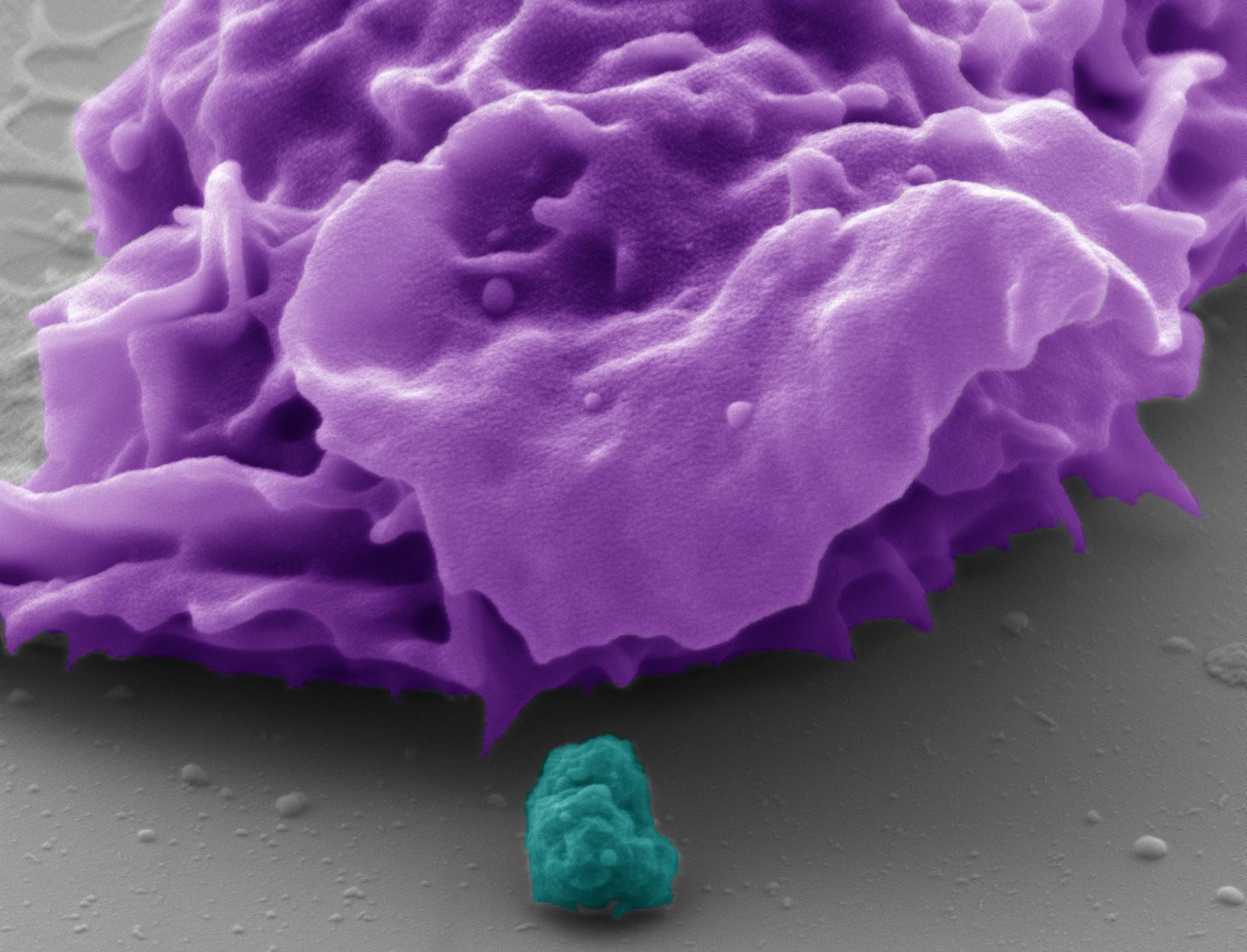
Wie professionelle Einbrecher brauchen auch bakterielle Krankheitserreger das passende Werkzeug, um unseren Körper zu besiedeln. Dabei sind einige Mikroben äußerste Spezialisten, die nur bestimmte Wirte befallen. Zu dieser kleinen Gruppe gehören die Gonokokken und der Erreger Haemophilus influenzae, die nur beim Menschen vorkommen. Beiden gemeinsam ist, dass sie geschickt verschiedene Abwehrmechanismen des menschlichen Körpers aushebeln, um sich auf der Schleimhaut festzusetzen.
Wie neue Arbeiten aus dem Labor von Prof. Dr. Christof Hauck, Zellbiologe an der Universität Konstanz, nun in der Fachzeitschrift „Current Biology“ zeigen, ist unser Körper auch diesen hochspezialisierten Bakterien nicht schutzlos ausgeliefert. Auf sogenannten Fresszellen unseres Immunsystems findet sich ein passendes Rezeptormolekül, das Erreger wie die Gonokokken erkennt und vernichtet. Überraschenderweise gibt es diesen hochspezialisierten Bakterienfänger, der CEACAM3 genannt wird, nur beim Menschen und seinen nächsten tierischen Verwandten wie dem Schimpansen, Gorilla oder Rhesusaffen: Jonas Adrian und Patrizia Bonsignore aus der Arbeitsgruppe von Christof Hauck untersuchten die Genome verschiedener Affenarten auf das Vorhandensein von CEACAM3. Sie konnten ein entsprechendes Rezeptormolekül nur bei höher entwickelten Affen, nicht jedoch bei Lemuren oder anderen „niederen“ Affen finden.
„Diese Erkenntnis deutet darauf hin, dass dieses Rezeptormolekül erst vor verhältnismäßig kurzer Zeit in der Evolutionsgeschichte der Primaten entstanden ist“, so Hauck. Beim Vergleich dieses Rezeptors zwischen verschiedenen Menschenaffen stellte sich zusätzlich heraus, dass sich CEACAM3 erstaunlich schnell weiterentwickelt. Zu erklären ist diese rasante Entwicklung durch die Funktion des Rezeptors als Abfangjäger für spezialisierte Bakterien: Jede Veränderung auf der Bakterienoberfläche, durch die der Erreger einen Vorteil gegenüber dem Immunsystem erzielt, zieht im Laufe von Generationen eine entsprechende Veränderung und Weiterentwicklung von CEACAM3 nach sich. „Entstanden ist auf diese Weise eine Art molekularer Rüstungswettlauf, bei dem manchmal die Mikroben, manchmal das Immunsystem die Nase vorn hat“, erklärt Christof Hauck.
Dass wir uns hier in einem laufenden Wettstreit befinden, zeigt der Blick auf die globale Vielfalt von CEACAM3. „In manchen menschlichen Populationen, beispielsweise auf dem afrikanischen Kontinent, existieren Varianten dieses Rezeptors. Dieser detaillierte Einblick in die Genome menschlicher Populationen ist erst durch die globale Datenerhebung der letzten Jahre möglich“, erläutert Jonas Adrian. Mit Blick auf die Funktion von CEACAM3 lag die Vermutung nahe, dass diese CEACAM3-Varianten ermöglichen, zusätzliche Erreger zu erkennen und zu eliminieren. Zur Prüfung dieser Vermutung führten Adrian und Bonsignore Bindungsstudien mit Varianten des Rezeptors und verschiedenen bakteriellen Krankheitserregern durch. Dabei werden die Rezeptormoleküle den in der menschlichen Population vorkommenden Varianten nachempfunden, im Labor hergestellt und gezielt mit den untersuchten Erregern in Kontakt gebracht, um die Bindung an die Erreger zu testen. In der Tat konnten die Wissenschaftler so zeigen, dass diese besonderen CEACAM3 Varianten zusätzliche Erreger erkennen, darunter Haemophilus influenzae. Während also die meisten Menschen mittels CEACAM3 spezialisierte Erreger eindämmen können, sind Personen mit einer CEACAM3 Variante sogar in der Lage, eine noch größere Bandbreite an Bakterien in Schach zu halten.
Der andauernde Wettstreit zwischen Mensch und Mikroben führt eindrücklich vor Augen, wie die Mechanismen der Evolution auch das menschliche Genom geformt haben. Dabei deutet die ungewöhnlich schnelle Entwicklung von CEACAM3 darauf hin, dass ein passgenauer Rezeptor vorteilhaft für unsere Primatenvorfahren war, um sich gegen hochspezialisierte Krankheitserreger zu behaupten.
Faktenübersicht:
- Originalpublikation: Adrian, J., Bonsignore, P., Hammer, S., Frickey, T., Hauck, C.R. (2019): Adaptation to host-specific bacterial pathogens drives rapid evolution of a human innate immune receptor, Current Biology 29, 1-15. https://doi.org/10.1016/j.cub.2019.01.058. Online publiziert am 7. Februar 2019
- Wettstreit zwischen menschlichem Immunsystem und bakteriellen Krankheitserregern
- Forschungsergebnisse weisen auf junge evolutionäre Entwicklung des Rezeptors CEACAM3 hin
- Die Erstautoren Jonas Adrian und Patrizia Bonsignore sind Mitglieder der Graduiertenschule Biological Sciences der Universität Konstanz
- Forschung in Kooperation mit Prof. Dr. Tancred Frickey (Scion, Neuseeland), vormals Juniorprofessor am Fachbereich Biologie der Universität Konstanz sowie der Graduiertenschule Chemische Biologie, die im Rahmen der Exzellenzinitiative bis 2019 gefördert wird
- Förderung durch die Deutsche Forschungsgemeinschaft
Presseinformation Nr. 14/2019
